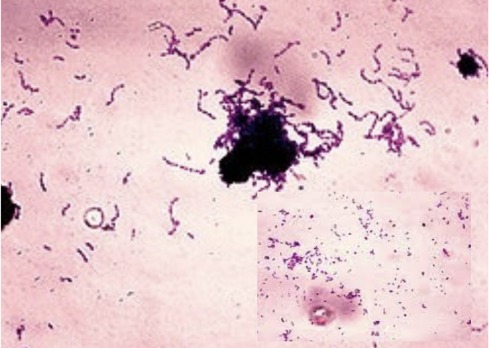
Um estudo da Universidade da Califórnia, em Berkeley, trouxe novas pistas sobre como bactérias da boca contribuem para o surgimento das cáries. A pesquisa, publicada em 19 de agosto na revista Proceedings of the National Academy of Sciences (PNAS), identificou um agrupamento genético capaz de favorecer a adesão de microrganismos aos dentes, fortalecendo a formação da placa bacteriana.
A investigação é liderada pela professora de Engenharia Química e Biomolecular, Li Zhang, que há anos se dedica a desvendar o microbioma oral. O objetivo de sua equipe é separar as bactérias consideradas benéficas das que estão associadas ao desenvolvimento de doenças dentárias, como a cárie, e encontrar formas de estimular as primeiras em detrimento das segundas.
O grupo de Berkeley analisou sequências de DNA de diversas bactérias presentes na boca – o chamado metagenoma – em busca de genes relacionados às lesões dentárias. Entre os achados, está um conjunto de cerca de 15 segmentos de DNA que produz duas moléculas com papéis complementares: uma funciona como “cola”, ajudando as células a se unirem, enquanto a outra age como um “fio”, permitindo a formação de cadeias. Juntas, elas favorecem a construção do biofilme resistente que recobre os dentes.
Esse agrupamento aparece em algumas cepas de microrganismos já conhecidos por sua relação com a cárie, como o Streptococcus mutans. A descoberta abre espaço para uma estratégia ousada: transferir esse mesmo mecanismo para bactérias consideradas benéficas, como a Streptococcus salivarius, usada hoje como probiótico oral, mas que ainda não consegue formar biofilmes duradouros.
“Certas cepas, da mesma espécie, podem ser patogênicas, comensais ou até probióticas”, explicou Zhang. “Se conseguirmos compreender melhor a atividade dessas moléculas, poderemos introduzi-las em bactérias ‘boas’, de forma que superem as produtoras de ácido que causam cáries.”
Metabolismo
A análise foi conduzida pela doutoranda McKenna Yao, que examinou uma base de dados com sequências metagenômicas de voluntários humanos. Após identificar agrupamentos genéticos suspeitos, a equipe cultivou bactérias em laboratório e isolou os metabolitos produzidos – pequenas moléculas compostas por peptídeos e lipídios.
De acordo com os pesquisadores, esse tipo de via genética é um exemplo de metabolismo “especializado”, que não é essencial para a sobrevivência da bactéria, mas pode alterar profundamente o ambiente onde ela vive. Em outras áreas da microbiologia, metabólitos desse tipo já se mostraram cruciais para a produção de antibióticos.
“Essas substâncias aumentam a sobrevivência bacteriana de várias formas”, explicou Yao. “Muitos atuam como antibióticos, eliminando rivais, enquanto outros ajudam na aquisição de metais, garantindo vantagens no ambiente.”
Implicações
O trabalho reforça a importância de explorar o papel dos metabólitos secundários no microbioma humano, ainda pouco estudado. Dois anos atrás, o mesmo grupo havia identificado um outro gene que produzia moléculas com efeito antibiótico. Agora, a descoberta das chamadas “mutanoclumpinas” pode oferecer novas armas contra as cáries.
Se estudos futuros confirmarem que essas moléculas estão diretamente relacionadas à doença, seria possível desenvolver inibidores genéticos ou químicos para impedir sua produção. Ao mesmo tempo, a equipe estuda formas de potencializar as bactérias benéficas, transformando-as em aliadas mais eficazes na proteção contra os microrganismos nocivos.
Apesar do avanço, Zhang e seus colegas fazem questão de lembrar que a escovação segue sendo a medida mais simples e eficiente para remover a placa bacteriana. “Ainda estamos começando a compreender a complexidade do que acontece dentro da boca”, destacou Yao.
O estudo contou com apoio do National Institute of Dental & Craniofacial Research, dos National Institutes of Health (NIH). Além de Zhang e Yao, assinam o artigo os pesquisadores Nicholas Zill, Colin Charles Barber, Yongle Du, Rui Zhai, Eunice Yoon e Dunya Al Marzooqi, todos de Berkeley, além de Peijun Lin, visitante da Faculdade de Computação, Ciência de Dados e Sociedade.